Climate science for biologists
(Scroll down for the German version)
I remember it well. Roughly 3 years ago, I was approached by Richard Davy – a climate scientist – to chat about what I was doing for my master’s thesis. At the time, I was conceptualising a way to calculate vegetation memory for global dryland regions. The resulting manuscript has been in submission for almost two years now, but that is a story for another time. When I told Richard I was planning to use WorldClim data for my environmental covariate data needs he looked confused. He hadn’t heard of WorldClim before. So, I explained to him what WorldClim was. Confusion in his eyes turned to disbelief before it settled on shock. He exclaimed: “Climate science hasn’t used data sets like these for around two decades now”. That is where my journey down the rabbit hole of climate science for use in biological research started. It hasn’t ended yet. Today, let me share some important resources with you, highlight some key findings, and indulge me as I celebrate the publication of my very first first-author paper.
Fig. 1. Erik Kusch and Richard Davy - the authors of KrigR | Erik Kusch und Richard Davy - Die KrigR-Autoren
Advances in climate science have rendered obsolete the gridded observation data widely used in downstream applications Such gridded observation products come in many shapes and sizes, but most familiar to us in the biological research sector will be WorldClim and CRU. Both data sets were major developments for macroecological research at the time – one could even say they were as revolutionary as the invention of the wheel. Unfortunately, as climate science progressed, biologically failed to keep up. Gridded observation products have turned into support wheels which many biologists are unprepared to shed thus infantilising our climate-driven large-scale research practices. What makes me say that? There are four important reasons why I strongly believe that the use of gridded observation products are actively holding us back.
- Data accuracy. Gridded observation data sets use observational data from individual weather stations. These are interpolated between station locations and harmonised to a pre-defined grid (usually 1x1km). No surprises here. However, what often goes unappreciated is that neither are weather stations distributed homogeneously across the Earth, nor are weather patterns smooth. Consequently, many parts of the Earth are terribly undersampled by weather station data alone. We require higher data accuracy.
- Temporal resolution. Most gridded observation products resolve weather events at monthly intervals. Obviously, this is insufficient for capturing extreme weather events at shorter duration and so doesn’t allow for quantification of exposure to extreme conditions. We require finer temporal resolution data.
- Data availability. Biological research has traditionally focussed on only two environmental variables and derivatives thereof: temperature and precipitation. An entire environment can hardly be condensed into just two variables. Setting aside that interpolation of precipitation data is difficult at best, for a science that prides itself on complexity, like biology, treating environment as such is disingenuous at best and wilfully neglectful at worst. We need more environmental variables.
- Data uncertainty. This goes hand in hand with data accuracy. Recognising that our data isn’t perfect is the first step to doing better research. Unfortunately, gridded observation data products don’t allow us to do so. Why? Because there are no quality indicators for the data points stored within them. We need measurements of data quality or uncertainty.
I hope that, by now, I have convinced you that using gridded observation data sets is doing yourself and our discipline a disfavour. So, what should we use instead? Are there any data sets that meet the requirements I outlined above? Yes. Climate Reanalyses.1
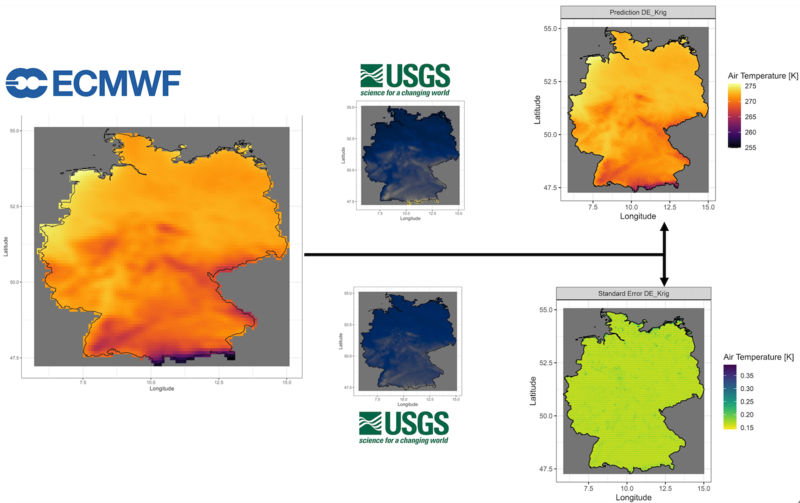
Fig 2. Data Pipeline with KrigR - (1) Obtain and preprocess data from ECMWF, (2) obtain and preprocess kriging covariate data (e.g. from USGS), and (3) statistically interpolate data using kriging methodology | Der Datrenprozess mit KrigR - (1) Herunterladen von Klimatdaten von ECMWF, (2) Herunterladen von Kriging-Ko-variatendaten (z.B.: von USGS), und (3) statistische Interpolation mit Kriging.
Climate Reanalyses are neat. No other way to say it. They incorporate far more than just weather station data thus being more accurate than gridded observation data products. They report data at hourly intervals! Global reanalyses data sets usually contain around 50-90 environmental variables with local reanalyses containing even more variables. Lastly, due to ensemble modelling, reanalyses data comes with uncertainty indicators. Consequently, climate reanalyses are the data products we ought to use. Yesterday at best.
Why haven’t climate reanalyses found wide-spread use in biology? I reckon we haven’t adopted their use yet for two major reasons:
- Climate reanalyses are more difficult to retrieve than gridded observation data sets and much larger in storage. Unless we have a straightforward, preferably R-contained, framework for data retrieval and handling of climate reanalyses, I fear biologists will keep relying on their support wheels.
- Spatial resolution of climate reanalyses data sets is coarser than gridded observation data sets. Biologists have gotten used to climate data sets at 1x1km resolution. Unfortunately, spatial resolution seems to have become king while data accuracy has been relegated to the position of a jester. I am personally incredibly dissatisfied with the fetishism for high spatial resolution at the cost of accuracy. However, I also realise that climate reanalyses will only find widespread use in biological research when we can obtain them at 1x1km resolutions.
Enter KrigR. KrigR is an R-package I have developed over the last two years to solve exactly these problems. KrigR provides an R-internal workflow for retrieval and handling of ERA5(-Land) – the world-leading global climate reanalysis data product. KrigR also comes with a statistical interpolation function which uses kriging – a geospatial interpolation method which allows for investigation of uncertainty values. With this tool, R-users can obtain state-of-the-art climate data tailored exactly to their study needs with as little as one function call.
To read more about this step-improvement for proficient use of climate data in biological research, please consult my very first first-author paper: https://doi.org/10.1088/1748-9326/ac48b3. There, I introduce the R-package and it’s use. For a more in-depth workshop of the capabilities of KrigR, please find extensive material on my personal website: https://www.erikkusch.com/post/krigr-mats/krigrworkshop/. Lastly, if you are still on the fence about just how much of a difference KrigR-obtained data makes when compared against traditional data sets, we have you covered. Look no further than https://doi.org/10.1088/1748-9326/ac39bf where Richard and I show you just how much of a discrepancy there is between state-of-the-art data and widely used climate data in biological research.
[GER] Klimawissenschaft für Biologen
Ich erinnere mich als wäre es gestern. Vor ungefähr 3 Jahren wurde ich von Richard Davy – einem Klimawissenschaftler – angesprochen, um über meine Masterarbeit zu sprechen. Zu dieser Zeit entwarf ich eine Methode zur Berechnung des Vegetationsgedächtnisses für globale Trockengebiete. Das entstandene Manuskript ist nun seit fast zwei Jahren eingereicht aber immer noch nicht publiziert aber das ist eine Geschichte für ein anderes Mal. Als ich Richard sagte, dass ich vorhabe, WorldClim-Daten für meine Umweltkovariatendaten zu verwenden, sah er verwirrt aus. Er hatte noch nie von WorldClim gehört. Also erklärte ich ihm, was WorldClim ist. Verwirrung in seinen Augen verwandelte sich in Unglauben, bevor es zu Schock wurde. Er rief: „Die Klimawissenschaft verwendet solche Datensätze seit etwa zwei Jahrzehnten nicht mehr.“ Hier begann meine Reise durch die Klimawissenschaft zur Verwendung in der biologischen Forschung. Die Reise ist noch lange nicht zu Ende. Im Folgenden erkläre ich wichtige Ressourcen, präsentiere wichtige Ergebnisse meiner Arbeit und zelebriere die Veröffentlichung meiner allerersten Erstautorenarbeit.
Fortschritte in der Klimawissenschaft haben die gerasterten Beobachtungsdaten, die in nachfolgenden Anwendungen weit verbreitet sind, obsolet gemacht. Solche gerasterten Beobachtungsprodukte gibt es in vielen Formen und Größen, aber im biologischen Forschungssektor werden uns WorldClim und CRU am bekanntesten sein. Beide Datensätze waren damals wichtige Entwicklungen für die makroökologische Forschung – man könnte sogar sagen, sie waren so revolutionär wie die Erfindung des Rades. Leider hielt die Biologie mit dem Fortschritt der nicht mit. Gerasterte Beobachtungsprodukte haben sich zu Stützrädern entwickelt, auf die viele Biologen immer noch angewiesen sind. Klimagetriebene, groß angelegten Forschungsprojekte werden dadurch infantilisiert. Was lässt mich das sagen? Es gibt vier Gründe, derentwegen ich fest davon überzeugt bin, dass uns die Verwendung von gerasterten Beobachtungsprodukten unsere Forschung aktiv verschlechtert.
- Datengenauigkeit. Gerasterte Beobachtungsdatensätze verwenden Beobachtungsdaten von einzelnen Wetterstationen. Diese werden zwischen Stationsstandorten interpoliert und auf ein vordefiniertes Raster (normalerweise 1x1km) abgestimmt. So weit, so erwartet. Was jedoch oft unbeachtet bleibt, ist, dass weder Wetterstationen homogen über die Erde verteilt sind noch Wettermuster uniform sind. Folglich sind weite Teile der Erde durch Wetterstationsdaten allein schrecklich schlecht repräsentiert. Wir brauchen bessere Datengenauigkeit.
- Zeitliche Auflösung. Die meisten gerasterten Beobachtungsprodukte repräsentieren Wetterereignisse in monatlichen Abständen. Dies reicht natürlich nicht aus, um extreme Wetterereignisse mit kürzerer Dauer zu erfassen, und ermöglicht daher keine Quantifizierung der Exposition gegenüber extremen Bedingungen. Wir benötigen feinere zeitliche Auflösung.
- Datenverfügbarkeit. Die biologische Forschung hat sich traditionell nur auf zwei Umweltvariablen und deren Derivate konzentriert: Temperatur und Niederschlag. Eine ganze Umgebung lässt sich kaum in nur zwei Variablen widerspiegeln. Abgesehen davon, dass die Interpolation von Niederschlagsdaten bestenfalls schwierig ist, ist die Behandlung der Umwelt als solche für eine Wissenschaft, die sich ihrer Komplexität rühmt, wie die Biologie, bestenfalls unaufrichtig und im schlimmsten Fall absichtlich nachlässig. Wir brauchen mehr Klimavariablen.
- Datenunsicherheit. Dies geht Hand in Hand mit der Datengenauigkeit. Zu erkennen, dass unsere Daten nicht perfekt sind, ist der erste Schritt zu besserer Forschung. Leider erlauben uns gerasterte Beobachtungsdatenprodukte dies nicht. Wieso das? Es gibt keine Qualitätsindikatoren für die darin gespeicherten Datenpunkte. Wir brauchen Messungen der Datenqualität oder Unsicherheit.
Ich hoffe, dich inzwischen davon überzeugt zu haben, dass die Verwendung von gerasterten Beobachtungsdatensätzen dir und unserer Disziplin schadet. Was sollten wir stattdessen verwenden? Gibt es Datensätze, die die oben beschriebenen Anforderungen erfüllen? Ja. Klima-Reanalysen.
Klima-Reanalysen sind genial. Anders kann man es nicht sagen. Sie beinhalten weit mehr als nur Wetterstationsdaten und sind daher genauer als gerasterte Beobachtungsdatenprodukte. Sie melden Daten im Stundentakt! Globale Reanalyse-Datensätze enthalten normalerweise etwa 50-90 Klimavariablen, wobei lokale Reanalysen noch mehr Variablen enthalten. Schließlich enthalten Reanalysedaten aufgrund der Ensemble-Modellierung Unsicherheitsindikatoren. Folglich sind Klimareanalysen die Datenprodukte, die wir verwenden sollten. Am besten schon Gestern.
Warum haben Klimareanalysen in der Biologie bisher keine breite Anwendung gefunden? Ich denke, wir haben ihre Verwendung aus zwei Hauptgründen noch nicht übernommen:
- Klimareanalysen sind schwieriger abzurufen als gerasterte Beobachtungsdatensätze und viel größer im Speicher. Ich fürchte, Biologen werden sich weiterhin auf ihre Stützräder verlassen, wenn wir keinen einfachen, vorzugsweise R-internen Prozesse für den Datenabruf und die Handhabung von Klima-Reanalysen haben.
- Die räumliche Auflösung von Klima-Reanalyse-Datensätzen ist gröber als gerasterte Beobachtungsdatensätze. Biologen haben sich an Klimadatensätze mit einer Auflösung von 1x1km gewöhnt. Leider scheint die räumliche Auflösung König geworden zu sein, während die Datengenauigkeit auf die Position eines Narren verbannt wurde. Ich persönlich bin unglaublich unzufrieden mit dem Fetischismus für hohe räumliche Auflösung auf Kosten der Genauigkeit. Mir ist jedoch auch klar, dass Klimareanalysen nur dann breite Anwendung in der biologischen Forschung finden werden, wenn wir sie mit einer Auflösung von 1x1 km erhalten.
Die Lösung? KrigR! KrigR ist ein R-Paket, das ich in den letzten zwei Jahren entwickelt habe, um genau diese Probleme zu lösen. KrigR bietet einen R-internen Workflow zum Abrufen und Bearbeiten von ERA5(-Land) – dem weltweit führenden globalen Klima-Reanalyse-Datenprodukt. KrigR verfügt auch über eine statistische Interpolationsfunktion, die Kriging verwendet – eine räumliche Interpolationsmethode, die die Untersuchung von Unsicherheitswerten ermöglicht. Mit diesem Tool können R-Anwender mit nur einem Funktionsaufruf Klimareanalysedaten abrufen, die exakt auf ihre Studienbedürfnisse zugeschnitten sind. Um mehr über diesen Schritt vorwärts für die kompetente Nutzung von Klimadaten in der biologischen Forschung zu erfahren, kann ich (ganz unvoreingenommen natürlich) meinen allerersten Erstautorenbeitrag zur Forschung empfehlen: https://doi.org/10.1088/1748-9326/ac48b3. Dort stelle ich das R-Paket und seine Verwendung vor. Für einen tiefergehenden Workshop zu den Fähigkeiten von KrigR findest du umfangreiches Material auf meiner persönlichen Website: https://www.erikkusch.com/post/krigr-mats/krigrworkshop/. Wenn du dir noch nicht sicher bist, welchen Unterschied KrigR-bezogene Daten im Vergleich zu herkömmlichen Datensätzen machen, habe ich auch was für dich. In https://doi.org/10.1088/1748-9326/ac39bf zeigen Richard und ich dir, wie groß die Diskrepanz zwischen State-of-the-Art-Daten und weit verbreiteten Klimadaten in der biologischen Forschung ist .



