Unleashing MOSAIC!
Unleashing MOSAIC, a database for interoperable macro-ecological research
[Versión en español más abajo]
In 2015, after a decade of work, we published an open-access database of plant demographic information (COMPADRE; Salguero-Gómez et al. 2015), and shortly after the animal counterpart, COMADRE (Salguero-Gómez et al. 2016). Common to COMPADRE and COMADRE, is the tool: matrix population models, where the life cycle of the species is organised along discrete stages (e.g. age, ontogeny, size ranges). More recently, we launched the PADRINO database (Levin et al. 2022), which contains information in the shape of Integral Projection Models, i.e. state predictors are continuous (e.g. size, volume, etc.). Collectively, these databases comprise the largest structured population dataset for comparative biodemography in the world.
Together, COMPADRE, COMADRE, and PADRINO cover 12,482 MPMs and 77 IPMs across 433 animal and 824 plant species and continue to grow. The sheer volume of high-resolution demographic information and pertinent metadata have allowed us and the wider research community to ask a wide range of ecological, evolutionary, and conservation biology questions. However, whenever we wanted to engage in large macro-ecological research, we encountered a challenge: interoperability.
The co-founder of the “www”, Sir Tim Berners-Lee, once famously said “I’m not interested in your data; I’m interested in merging your data with other data. Your data will never be as exciting as what I can merge it with”. What Berners-Lee was referring to is interoperability: the ability of open-access resources to “speak” to each other so that their product is more than the mere summation of their elements. In practice, this means building an ecosystem of data structures that communicate with each another through standardised, linked attributes, such as populations or species, and supporting the scaling of data from individuals to populations, species, or higher order taxonomic or functional groupings.
For quite a few years, we have aspired to complement COMPADRE, COMADRE, and PADRINO with data that do not fall exactly within their niche of operation (i.e. demography), but that would greatly expand the range of questions they can be used for. We periodically found ourselves having to use databases that (at least up until recently) were not fully open access, and as such, we had to put in the hard work to find information regarding, e.g., species mobility, mode of parity, growth form, etc.
To facilitate this task to users wanting to carry out research with COMPADRE, COMADRE, and PADRINO, we introduce MOSAIC. MOSAIC, whose name reflects the wide heterogeneity of complementary data there in, including biomass, dispersal mode, mating systems, and volancy (flight capability. In addition to a wide spectrum of continuously and discrete traits, MOSAIC compiles state-of-the-art climate data from ERA-5 Land with the ECMWF is distributed through MOSAIC, providing for the first time, immediate access to climate variables for all populations modelled in COMADRE, COMPADRE, and PADRINO. MOSAIC is also centralises a phylogenetic tree for species within the structured population databases, drawn from Online Tree of Life, streamlining access to phylogenetic data to account for nonindependence in analyses. MOSAIC is a catch-all database to augment population models with information on species traits and their biotic and abiotic environments, and is slated to grow alongside COMADRE, COMPADRE, and PADRINO.
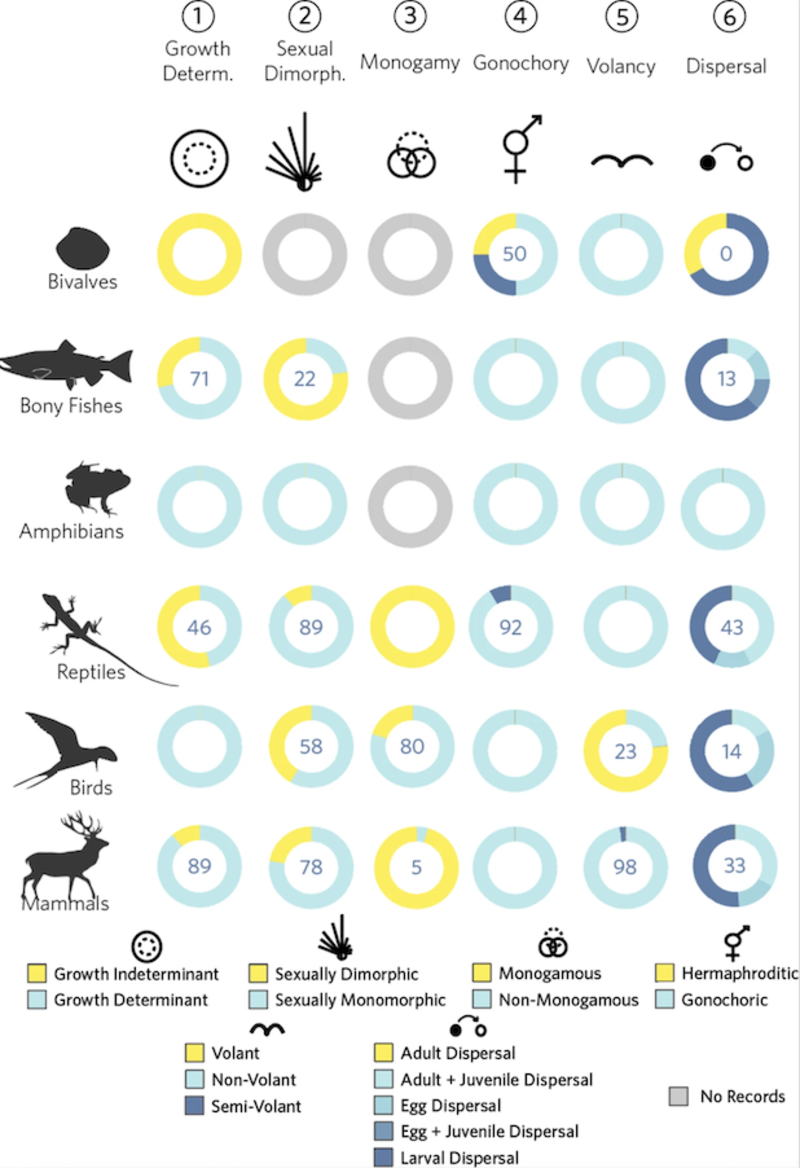
Fig. 1. Representation of the data currently contained in MOSAIC. [SPA] Representación de los datos actualmente contends en MOSAIC.
Our hope is that by unleashing this database open-access too (and making R scripts fully available), users will sail the waters of macroecology in better company. The database can be found at https://mosaicdatabase.web.ox.ac.uk and the publication introducing it just appeared in Scientific Data (https://www.nature.com/articles/s41597-023-02070-w). Go enjoy it and explore interesting questions/hypotheses!
Written by Connor Bernard and Rob Salguero-Gomez
[Versión en español]
En 2015, tras una década de trabajo, publicamos una base de datos de acceso totalmente abierto que contine información demográfica del reino vegetal (COMPADRE; Salguero-Gómez et al. 2015), y poco después la contraparte animal, COMADRE (Salguero-Gómez et al. 2016). El denominador común entre COMPADRE y COMADRE es la herramienta: modelos matriciales de población (MPM por sus singlas en inglés), en los cuales el ciclo de vida de las especies se organiza a lo largo de etapas discretas (por ejemplo, edad, ontogenia, rangos de tamaño). Más recientemente, lanzamos la base de datos PADRINO (Levin et al. 2022), la cual contiene información en forma de modelos de proyección integral (IPM por sus siglas en inglés), es decir, los predictores de estado son continuos (por ejemplo, tamaño, volumen, etc.). Colectivamente, estas bases de datos contienen el mayor conjunto de datos de población estructurada para biodemografía comparativa.
Juntos, COMPADRE, COMADRE y PADRINO cubren 12,482 MPMs y 77 IPMs en 433 animales y 824 especies de plantas… y continúan creciendo. El gran volumen de información demográfica de alta resolución y los pertinentes metadatos nos han permitido a nuestro grupo de investación y a la comunidad investigadora en general estudiar una amplia gama de preguntas ecológicas, evolutivas y de biología de la conservación. Sin embargo, cada vez que hemos querido participar en una investigación macroecológica a gran escala, nos hemos encontrado con un importante desafío: la interoperabilidad.
El cofundador de “www”, Sir Tim Berners-Lee, dijo una vez: “No estoy interesado en tus datos; Estoy interesado en fusionar tus datos con otros datos. Tus datos nunca serán tan interesantes como aquellos con los que puedo fusionarlos”. A lo que se refería Berners-Lee es a la interoperabilidad: la capacidad de los recursos de acceso abierto de “hablar” entre sí para que su producto sea más que la mera suma de sus elementos. En la práctica, ello significa construir un ecosistema de estructuras de datos que se comunican entre sí a través de atributos vinculados y estandarizados, como poblaciones o especies, y respaldar la escala de datos de individuos a poblaciones, especies o agrupaciones funcionales o taxonómicas de orden superior.
Durante bastantes años, hemos aspirado a complementar COMPADRE, COMADRE y PADRINO con datos que no caen exactamente dentro de sus nichos de operación (es decir, la demografía), pero que ampliarían mucho el rango de preguntas para las que pueden ser utilizados. Periódicamente, nos hemos vimos obligados a usar bases de datos que (al menos hasta hace poco) no eran de acceso totalmente abierto y, como tal, tuvimos que trabajar duro para encontrar información sobre, por ejemplo, la movilidad de las especies, el modo de paridad, la forma de crecimiento, etc.
Para facilitar esta arduosa tarea a los usuarios que quieran realizar investigaciones con COMPADRE, COMADRE y PADRINO, hoy presentamos la nueva base de datos MOSAIC. El nombre MOSAIC refleja la amplia heterogeneidad de datos complementarios que contiene, incluyendo la biomasa, el modo de dispersión, los sistemas de apareamiento, la capacidad de volar, etc.. Además de un amplio espectro de rasgos continuos y discretos, MOSAIC compila Los datos climáticos de ERA-5 Land con el ECMWF se distribuyen a través de MOSAIC, proporcionando por primera vez, acceso inmediato a las variables climáticas para todas las poblaciones modeladas en COMADRE, COMPADRE y PADRINO. MOSAIC también contiene un árbol filogenético para especies de las bases de datos demograficas, extraído de Online Tree of Life, por ende simplificando el acceso a los datos filogenéticos para tener en cuenta la falta de independencia en los análisis comparativos.
Nuestra esperanza es que, al hacer “open-access” esta base de datos, asi como los pertinentes scripts de R, los usuarios navegarán las aguas de la macroecología en mejor compañía. La base de datos está contenida en https://mosaicdatabase.web.ox.ac.uk y la publicación que la presenta acaba de aparecer en Scientific Data (https://www.nature.com/articles/s41597-023-02070-w) . ¡A disfrútarla y explorar preguntas/hipótesis interesantes!
Escrito por Connor Bernard y Rob Salguero-Gómez, y traducido por Rob Salguero-Gómez



